Gennaio 3, 2022 Grafene, Studi scientifici
Questi fori rivestono un ruolo di notevole importanza nell’interazione con altre sequenze di reticoli di origami di DNA, che presentano la possibilità di legami a formazione di un pezzo di Lego.
Prove di auto-assemblaggio dei reticoli di origami di DNA
Uno degli aspetti più complessi da determinare nel riconoscimento di pattern nei cosiddetti “vaccini” riguarda l’identificazione della procedura mediante cui gli oggetti osservati sperimentalmente (micro/nano-router, micro/nano-rectenne) sono in grado di assemblarsi.
La revisione della letteratura scientifica ha rivelato numerosi studi che illustrano diverse tecniche di assemblaggio, tra cui la litografia elettronica, il fascio ionico focalizzato FIB (Focused Ion Beam) e persino modelli sintetici di DNA utilizzati per definire i circuiti QCA.
Tuttavia, nei campioni dei cosiddetti “vaccini” non è stata trovata alcuna chiara evidenza di autoassemblaggio.
Tuttavia, si osserva che queste particelle sembrano aggregarsi per formare strutture più complesse, delineando schemi geometrici semplici.
In ambito scientifico, tale comportamento o movimento quasi diretto delle particelle, nell’ambito della costruzione di oggetti e dispositivi micro/nano elettronici in una nanorete di comunicazioni intracorporee, è altamente probabile che corrisponda a un processo di auto-assemblaggio basato sul DNA, crescita epitassiale ed origami.
Questa conclusione ha condotto alla localizzazione di articoli scientifici che, con elevata probabilità, potrebbero confermare l’auto-assemblaggio di oggetti complessi, inclusi circuiti, schede, router, sensori e altri componenti e dispositivi micro/nano elettronici.
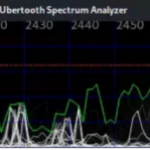
La presente indagine consente di comprendere come si verifichi l’auto-assemblaggio dei componenti responsabili del fenomeno di trasmissione dell’indirizzo MAC bluetooth (Sarlangue, G.; Devilleger, J.; Trillaud, P.; Fouchet, S.; Taillasson, L.; Catteau, G. 2021), quali micro/nano-router, micro/nano-antenne, micro/nano-rectenne, con cui si configura l’hardware della rete di nano-comunicazioni intra-corporea.
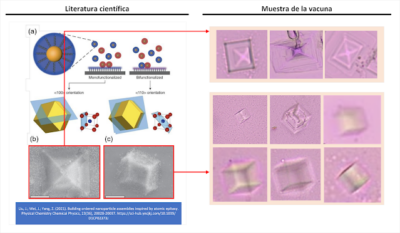
La Figura 1 illustra esempi di auto-assemblaggio osservati nella literatura scientifica e la loro corrispondenza con i campioni analizzati del cosiddetto “vaccino” Pfizer.
Dal punto di vista morfologico, si evidenziano significative coincidenze che consentono di inferire, con una probabilità elevata, che l’auto-assemblaggio possa costituire una realtà dimostrabile.

In considerazione della complessità dell’auto-assemblaggio e della rilevanza delle prove scoperte, si procederà con un’analisi dettagliata che si concentrerà su tre temi principali : a) auto-assemblaggio diretto ; b) auto-assemblaggio mediante crescita epitassiale regolare ; c) auto-assemblaggio di origami.
Auto-assemblaggio diretto
L’articolo di Kumar (2010) fornisce la prima evidenza chiara dell’autoassemblaggio diretto osservabile nel campione del cosiddetto “vaccino“, come illustrato in figura 2.
Le nanoparticelle osservate sembrano aggregarsi in cluster di dimensioni maggiori e successivamente in strutture più complesse che si muovono all’interno della goccia del campione.

Secondo l’opinione di Kumar (P. 2010), l’auto-assemblaggio diretto riveste un ruolo cruciale nello sviluppo di dispositivi elettronici, magnetici ed ottici miniaturizzati, che trovano applicazione nei materiali derivati dal grafene rinvenuti nei campioni del cosiddetto “vaccino”.
Infatti, l’autore sottolinea che “le nanoparticelle hanno attirato molta attenzione a causa delle loro proprietà uniche dipendenti dalle dimensioni, tra cui superparamagnetismo, chemiluminescenza e catalisi.
Per sfruttare appieno il potenziale delle nanoparticelle, è necessario sviluppare nuovi metodi per assemblare tali componenti in modelli o strutture utili.
Queste strutture auto-assemblanti offrono promettenti opportunità per lo sviluppo di dispositivi ottici, elettronici, optoelettronici e magnetici miniaturizzati“.
Inoltre, Kumar sottolinea che il metodo di “auto-assemblaggio diretto” è particolarmente adatto per la generazione di dispositivi su scala nano e micro, grazie alla sua capacità di utilizzare punti quantici o nanopunti.
Questo concetto è ulteriormente approfondito : “Man mano che le dimensioni o le funzionalità dei dispositivi si riducono, i processi litografici convenzionali diventano limitati per la loro produzione.
Si rende pertanto necessario lo sviluppo di metodi alternativi per superare tale criticità.
L’evoluzione delle tecnologie di produzione convenzionali, come la litografia ottica, si scontra con limiti fondamentali.Inoltre, si rende necessario lo sviluppo di nuove tecniche di produzione per prolungare la durata e la gamma di applicazioni delle tecniche esistenti.
La tecnica di auto-assemblaggio diretto può essere impiegata per la produzione di nanostrutture funzionali, quali nanofili e una serie organizzata di nano-punti (punti quantici)“.
In altre parole, l’auto-assemblaggio diretto consente ai GQD (Graphene Quantum Dots o punti quantici di grafene) di auto-assemblarsi secondo un modello predefinito.
Tra i diversi tipi di auto-assemblaggio diretto o guidato, Kumar considera “l‘assemblaggio guidato da modelli di superficie atomica ; l’assemblaggio guidato da un campo elettromagnetico o da un campo elettrico, da un fascio di elettroni, luce e laser, tra gli altri“.
Inoltre, l’autore sottolinea che “l’auto-assemblaggio diretto è una tecnica valida e facilmente riproducibile, con prospettive future per l’uso su scala industriale, che consente di costruire strutture ben ordinate, spesso di notevole interesse, e che presentano un’elevata facilità di organizzazione dei materiali su scala nanometrica, permettendo la produzione di strutture complesse su larga scala“.
Questo aspetto sembra assumere particolare rilevanza nel contesto della rete intracorporea di nanocomunicazioni e dei nano/micro dispositivi, in quanto è necessario creare un gran numero di dispositivi per garantire il corretto funzionamento dell’intero sistema (Zhang, R.; Yang, K.; Abbasi, QH; Qaraqe, KA; Alomany, A. 2017 | Galal, A.; Hesselbach, X. 2018 | Galal, A.; Hesselbach, X. 2020)
Tra tutte le forme di auto-assemblaggio, la più probabile e morfologicamente più coerente è l’auto-assemblaggio determinato da modelli di DNA biologico.
Tra i benefici associati, Kumar sottolinea la produzione di nanofili, che consente di risolvere problemi di integrazione (eliminando la necessità di manipolare singoli nanofili) e problemi di contatto per il trasporto elettrico e magnetico.
Questo concetto trova applicazione nei diversi tipi di nanodispositivi osservati, quali micro/nano retto e materiali derivati dal grafene, inclusi i punti quantici di grafene (GQD).
Inoltre, Kumar sostiene che “l’uso di modelli fisici di DNA porta alla crescita di nanomateriali in una posizione predefinita, eliminando la necessità di manipolazione post-crescita e fornendo la possibilità di collegamenti elettrici per ulteriori caratterizzazioni” (Kumar, 2023).
Questa affermazione aiuta a comprendere come le forme quadrangolari osservate nei campioni del cosiddetto “vaccino” siano costruite e definite, con una forte somiglianza con PCB (Printed Circuit Boards, cioè “schede a circuiti stampati“), microchip, sensori e circuiti integrati.
Inoltre, si osserva che tali modelli portano alla formazione di nanopunti (quantum dots), ovvero nanofili verticali che possono essere impiegati in modo controllato per la produzione di dispositivi FET (Field Effect Transistor), dispositivi di giunzione a tunnel magnetici e dispositivi per applicazioni ottiche.
Questo dimostra che, attraverso l’auto-assemblaggio diretto, è possibile creare nanotecnologie miniaturizzate per qualsiasi dispositivo elettronico noto.
In altre parole, l’auto-assemblaggio guidato da modelli di DNA biologico può essere utilizzato per realizzare tutti i dispositivi necessari per una nanorete intracorporea, e questa è probabilmente la tecnica utilizzata nei cosiddetti “vaccini“, come dimostrato sulla base delle immagini osservate e le dichiarazioni nella literatura scientifica (Catania, V.; Mineo, A.; Monteleone, S.; Patti, D. 2014 | Keren, K.; Berman, RS; Buchstab, E.; Sivan, U.; Braun, E. 2003).

Inoltre, Kumar osserva che “le strategie dirette dalle biomolecole (modelli biologici di DNA) hanno mostrato metodi altamente promettenti nell’assemblaggio delle nanoparticelle in un’ampia varietà di architetture“, grazie alla loro elevata efficienza, specifica e programmabilità genetica (McMillan, RA; Paavola, CD; Howard, J.; Chan, SL; Zaluzec, NJ; Trent, JD 2002).
Questi materiali nanoassemblati hanno dimostrato di avere potenziali applicazioni in nuovi sistemi di rilevamento, come i biosensori (Taton, TA; Mirkin, CA; Letsinger, RL 2000) e sensori chimici (Liu, J.; Lu, Y. 2003 | Liu, J.; Lu, Y. 2006), e nella costruzione di dispositivi nanoelettronici (Keren, K.; Berman, RS; Buchstab, E.; Sivan, U.; Braun, E. 2003) [paradossalmente configurati con nanotubi di carbonio].
Questo risultato consolida ulteriormente la convinzione che tale tecnica o metodo rappresenti un’opzione estremamente vantaggiosa per l’implementazione della nanotecnologia nel contesto del corpo umano.
Auto-assemblaggio tramite crescita epitassiale regolare
L’ipotesi dell’auto-assemblaggio diretto può essere considerata ben fondata, tuttavia l’auto-assemblaggio mediante crescita epitassiale regolare offre prove ancora più convincenti.
La Figura 4 illustra un’esatta corrispondenza tra la literatura scientifica e i campioni del cosiddetto “vaccino” Pfizer esaminati dal dott. Campra (Campra, P. 2021).
In particolare, alcuni elementi di forma quadrangolare e piramidale sembrano essere il risultato di una tecnica di auto-assemblaggio epitassiale, che a sua volta è “uno dei processi di fabbricazione dei circuiti integrati” (Shen, J. ; Sun, W. ; Liu, D. ; Schaus, T.Yin, P. 2021 | Burns, MA; Mastrangelo, CH; Sammarco, TS; Man, FP; Webster, JR; Johnsons, BN; Burke, DT 1996 | Esener, SC; Hartmann, DM; Heller, MJ; Cable, JM 1998 | Krahne, R.; Yacoby, A.; Shtrikman, H.; Bar-Joseph, I.; Dadosh, T.; Sperling, J. 2002 | Chen, Y.; Pepin, A. 2001).
L’epitassia è il processo di deposito di uno strato di materiale (ad esempio punti quantici di grafene, ossido di grafene, idrogel, ecc.) su di un substrato di nucleazione primaria.
Tuttavia, a differenza dei processi di crescita tradizionali, in questo caso si ottiene tramite ibridazione del DNA.
È in questa fase che (Liu, J.; Wei, J.; Yang, Z. 2021) sviluppano uno degli argomenti centrali della loro ricerca.
Secondo l’opinione di Liu, J. e Wei, J. (2021), l’auto-assemblaggio di “nanoparticelle inorganiche in assemblaggi di nanoparticelle mesoscopiche o macroscopiche rappresenta una strategia efficace per la fabbricazione di dispositivi avanzati con funzionalità emergenti su scala nanometrica.
Inoltre, l’assemblaggio di nanoparticelle su substrati può consentire la fabbricazione di dispositivi integrati, simile alla crescita di cristalli atomici.
I recenti progressi nel campo dell’assemblaggio di nanoparticelle suggeriscono che gli assemblaggi ordinati di nanoparticelle potrebbero essere ben prodotti su di un substrato selezionato, noto come crescita epitassiale morbida“.
La definizione di “epitassiale” indica una crescita controllata di cristalli su un substrato o uno stampo di DNA, che può essere utilizzata per la fabbricazione di dispositivi elettronici micro/nano.
Questo concetto è illustrato nel seguente modo : “l’ibridazione del DNA è stata applicata per assemblare nanoparticelle in superreticoli con strutture cristalline sorprendentemente ricche.
La struttura tridimensionale a doppia elica del DNA (passo fisso, diametro fisso) è risultata avere più vantaggi rispetto ad altri materiali nel guidare le nanoparticelle verso un assemblaggio tridimensionale ordinato” (Nykypanchuk, D.; Maye, MM; Van-Der-Lelie, D.; Gang, O. 2008).
Il riconoscimento specifico tra le coppie di basi, la capacità di controllare la lunghezza del filamento di DNA e la sequenza di basi ne fanno un strumento potente per l’assemblaggio su scala nanometrica.
La programmazione del DNA conferisce a quest’ultimo un elevato potenziale come legante orientato alla struttura.
La ricerca in questo ambito ha dimostrato che l’auto-assemblaggio del DNA non solo è in grado di generare strutture bidimensionali (2D), ma anche strutture tridimensionali (3D), grazie alla formazione di legami a doppia elica.
Questo apre la possibilità di utilizzare il DNA per la configurazione di una vasta gamma di forme, tra cui quelle cubiche e prismatiche, come evidenziato nella figura 4.
Tra gli studi citati da (Liu, J. ; Wei, J. ; Yang, Z., 2021) vi è un paragrafo sull’auto-assemblaggio epitassiale, che rivela una vasta esperienza nella sperimentazione di costrutti cristallini basati sul DNA, con una tolleranza di errore (mismatch) solo dell’1%.
Secondo Lewis, DJ; Zornberg, LZ; Carter, DJ; Macfarlane, RJ (2020), tale tecnica rappresenta una combinazione di nanoparticelle funzionalizzate con DNA e un filamento di DNA funzionalizzato con substrato, finalizzata alla progettazione di un processo di assemblaggio epitassiale.
In tale ambito, è stato rilevato che le forme Winterbottom a cristallo singolo di cristalli di nanoparticelle si formano mediante il controllo delle energie interfacciali tra i cristalli e il fluido, il substrato e il cristallo, ed il substrato e il fluido.
Altri esempi mostrano che le nanoparticelle innestate di DNA auto-assemblate in film colloidali bidimensionali possono essere applicate come substrato per un assemblaggio epitassiale morbido.
Ad esempio, (Wang, MX; Seo, SE; Gabrys, PA; Fleischman, D.; Lee, B.; Kim, Y.; Mirkin, CA 2017) hanno utilizzato nanoparticelle rivestite di DNA come elementi di costruzione più elastici e malleabili per adattarsi meglio alla mancata corrispondenza della rete.
Studi successivi (Gabrys, PA; Seo, SE; Wang, MX; Oh, E.; Macfarlane, RJ; Mirkin, CA 2018) hanno mostrato che i film sottili di super-reticolo assemblati da nanoparticelle funzionalizzate di DNA possono immagazzinare ceppi elastici deformandosi e riorganizzandosi, con disallineamenti del reticolo fino a ± 7,7%, superando significativamente i disallineamenti del reticolo di ± 1% consentiti dai film sottili atomici.
È fondamentale sottolineare che le nanoparticelle rivestite di DNA sperimentano un rilassamento progressivo e coerente, dissipando la deformazione in modo elastico ed irrecuperabile attraverso la formazione di dislocazioni o vuoti.
Pertanto, “è possibile sviluppare film colloidali eteroepitassiali controllando equivalenti atomici-soft-programmabili di nanometri e microstrutture utilizzando nanocristalli rigidi rivestiti con materiali polimerici morbidi e comprimibili“. (Liu, J.; Wei, J.; Yang, Z. 2021)
Origami auto-assemblanti
In ambito scientifico, tra le forme più originali di auto-assemblaggio si distingue il “metodo dell’origami“, anch’esso correlato all’impiego di modelli di DNA.
La prova di tale affermazione si rinviene nell’opera di (Wang, J. ; Yue, L. ; Li, Z. ; Zhang, J. ; Tian, H. ; Willner, I., 2019) dal titolo “Active generation of nano-holes in DNA origami scaffolds for programmed catalysis in nanocavities“.
Il modello di un punto o di un foro all’interno di una struttura quadrangolare è sorprendente e caratterizzante da un punto di vista morfologico.
Questo dettaglio è stato osservato nelle immagini acquisite dal Dr. Campra, che, in collaborazione con l’oggetto di studio autoassemblante, ha permesso di dedurre che si trattava di un elemento mancante e che, in realtà, devono essere presenti oggetti di maggiori dimensioni auto-assemblati mediante il metodo dell’origami.
Le somiglianze risultano evidenti, come evidenziato in figura 5, in cui la struttura quadrangolare degli oggetti, la posizione dei nano-fori inscritti all’interno della superficie, nonché il numero o la quantità osservati nei campioni del cosiddetto “vaccino” Pfizer, coincidono.
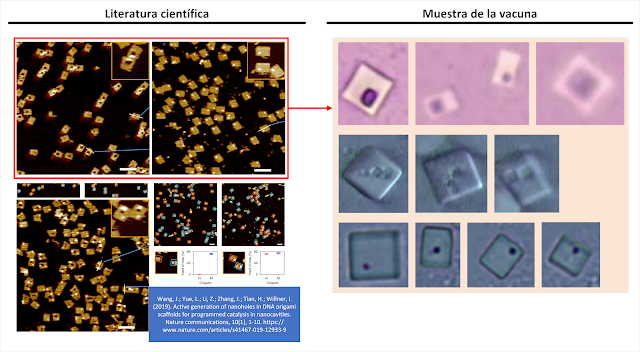
Prima di procedere con l’analisi dettagliata dei fori presenti negli oggetti quadrangolari, è opportuno rivedere l’introduzione e lo stato dell’arte fornito dagli autori nel loro articolo.
Questo aiuta a collocare le capacità della tecnica nel contesto appropriato e a dimostrarne il legame con la nanotecnologia utilizzata nei cosiddetti “vaccini“.
È interessante notare come l’auto-assemblaggio origami venga definito come un “assemblaggio programmato di nanostrutture di DNA bidimensionali (2D) e tridimensionali (3D), il che rappresenta un importante progresso nella nanotecnologia del DNA” (Hong, F.; Zhang, F.; Liu, Y.; Yan, H. 2017 | Rothemund, PW 2006 | Endo, M.; Sugiyama, H. 2014), confermando non solo le possibili dimensioni o gli assi dell’auto-assemblaggio, ma anche la compatibilità del metodo dell’origami con l’auto-assemblaggio con crescita epiteliale regolare, sia con l’auto-assemblaggio diretto che con quello guidato.
In ogni caso, l’impiego di strutture di DNA sintetico opportunamente configurate rappresenta i precursori necessari per lo sviluppo delle strutture e degli oggetti osservati nei campioni del cosiddetto “vaccino“.
Inoltre, (Wang, J. ; Yue, L. ; Li, Z. ; Zhang, J. ; Tian, H. ; Willner, I. 2019) confermano che il metodo di auto-assemblaggio dell’origami utilizzando il DNA consente l’ancoraggio di componenti per configurare, tra gli altri dispositivi, antenne plasmoniche, già in precedenza individuate nei campioni del cosiddetto “vaccino” come parte della nano-rete centrata sul corpo umano.
Un concetto espresso chiaramente nella seguente citazione : “Oltre a creare forme ingegnose di strutture di origami generate dal ripiegamento programmato del DNA, le strutture di origami sono state funzionalizzate con filamenti di acido nucleico sporgenti o filamenti di oligonucleotidi con bordi modificati.
I filamenti sporgenti sono stati impiegati come punti di ancoraggio per l’organizzazione di polimeri, proteine e nanoparticelle negli scaffold di ciascun origami.
Sono state dimostrate funzioni uniche di nanostrutture assemblate in scaffold di origami, tra cui il funzionamento di cascate di enzimi, la progettazione di antenne plasmoniche e l’assemblaggio di strutture chiroplasmoniche“.
Questa spiegazione è fondamentale per comprendere il processo di formazione delle sovrastrutture, guidate da pattern di DNA, in quanto sono collegate attraverso i filamenti sporgenti dei blocchi di costruzione, funzionalizzati con nanoparticelle (ad esempio, punti quantici di grafene GQD), che insieme al fattore di scala e superconduttore del materiale, forniscono caratteristiche plasmoniche e di hall quantistico, implicando l’auto-assemblaggio di transistor e micro/nano chip della complessità richiesta.
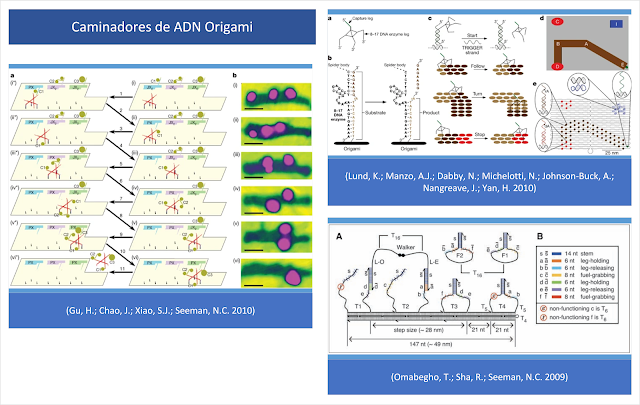
Nella loro introduzione, (Wang, J.; Yue, L.; Li, Z.; Zhang, J.; Tian, H.; Willner, I. 2019) forniscono anche annotazioni e citazioni di notevole interesse riguardo alle possibilità offerte dalla tecnica origami e dalla progettazione di DNA walkers con capacità motorie, in grado di iniziare il movimento, girarsi e fermarsi, secondo i modelli di interazione molecolare.
In effetti, secondo Lund et al. (2010), questi DNA walker rappresentano essenzialmente robot molecolari guidati da molecole di substrato (precursori) all’interno di strutture di DNA origami (modelli).
Questo concetto è ulteriormente rafforzato dalle seguenti citazioni di Lund, avvalorate anche da Omabegho, T. e Sha, R. (2009) e da Gu, H., Chao, J. e Seeman, NC (2010), i quali confermano che i DNA walker sono robot molecolari guidati da molecole di substrato (precursori) in un insieme di strutture di DNA origami (modelli).
“Trasferire la robotica al livello di una singola molecola è teoricamente fattibile, ma richiede di affrontare la capacità limitata delle singole molecole di immagazzinare informazioni e programmi complessi.
Una strategia per superare tale limite consiste nell’impiegare sistemi capaci di ottenere comportamenti complessi dall’interazione di semplici robot con il loro ambiente.
Un primo passo in questa direzione è stato compiuto con lo sviluppo dei cosiddetti DNA-walker, che sono passati dall’essere non autonomi alla capacità di eseguire movimenti brevi ma diretti su binari unidimensionali“.
In questa ricerca, si dimostra che i walker casuali, noti anche come “ragni molecolari“, composti da una molecola di streptavidina come corpo inerte e tre desossiribozimi come zampe catalitiche, manifestano un comportamento robotico elementare quando interagiscono con un ambiente definito con precisione.
Le osservazioni al microscopio a singola molecola confermano che questi camminatori raggiungono il movimento direzionale rilevando e modificando le tracce delle molecole di substrato disposte in un paesaggio di origami di DNA bidimensionale (Lund, K.; Manzo, AJ; Dabby, N.; Michelotti, N.; Johnson-Buck, A.; Nangreave, J.; Yan, H., 2010).
Questa affermazione può confermare la presenza di molecole e frammenti con la capacità di auto-assemblarsi, il loro movimento, orientamento e auto-organizzazione, e di configurare dispositivi elettronici complessi, secondo schemi e modelli di DNA sintetico.
Nel proseguire l’analisi di (Wang, J.; Yue, L.; Li, Z.; Zhang, J.; Tian, H.; Willner, I., 2019), si evidenzia che “la funzionalizzazione dei bordi di mosaici origami (di modelli di DNA) è stata applicata per progettare strutture origami multicomponenti programmate, con un focus particolare sullo sviluppo di dimeri origami intercambiabili“.
In altre parole, i modelli di DNA possono essere definiti in modo tale da essere composti da parti specifiche (particelle, proteine, punti quantici, ecc.) secondo un programma o un modello predeterminato.
La tecnologia origami del DNA può estendersi a ulteriori ambiti, come evidenziato dagli studi pionieristici condotti da Wang e dal suo team : “Sono stati sviluppati sistemi origami 3D ingegnosi.
Ad esempio, è stato osservato l’auto-assemblaggio di una scatola origami, l’assemblaggio graduale di strutture di DNA programmabili su scala gigadalton e il movimento guidato dalla luce di pacchetti origami 3D per produrre funzioni chiroptiche reversibili.
Sono state avanzate diverse proposte per le applicazioni delle nanostrutture di origami, tra cui catalisi programmata, rilascio controllato di farmaci, operazioni di gate logico e rilevamento“.
Tra le applicazioni menzionate, si evidenziano le operazioni di gate logico e rilevamento, caratteristiche della progettazione di circuiti QCA (Quantum Cell Automata), già menzionate nell’identificazione dei nanorouter tra i pattern osservati in precedenza nei cosiddetti “vaccini“.
Questo dimostra ulteriormente che la metodologia dell’origami del DNA è efficace per lo sviluppo di dispositivi elettronici basati su punti quantici, poiché consente di controllare la costruzione ordinata di cavi e circuiti.
Una volta completata la rassegna dei preamboli all’articolo di (Wang, J.; Yue, L.; Li, Z.; Zhang, J.; Tian, H.; Willner, I. 2019), il discorso scientifico si concentra sull’oggetto delle cavità o fori nei cosiddetti “origami rafts“, che nell’inquadratura sono mostrati come strutture quadrangolari con un punto inscritto nella loro area.
Gli autori dello studio hanno affermato che “la maggior parte di queste strutture di origami funzionali implica la modifica dal basso verso l’alto di zattere di origami, la modifica dei bordi delle tessere di origami o il piegamento delle tessere in tubi.
Tuttavia, si può prendere in considerazione la funzionalizzazione di strutture di origami con nanocavità (fori o barili) che potrebbero comportarsi da contenimento o da canali per trasformazioni chimiche guidate.
Finora, tali cavità sono state prodotte durante l’assemblaggio passivo di piastrelle origami e sono state impiegate per il docking sito-specifico di anticorpi, la ricostituzione di proteine di membrana e la funzionalizzazione di pori a stato solido per il trasporto selettivo.
Inoltre, si è osservata l’introduzione di strutture di DNA (non origami) all’interno delle membrane, le quali hanno funzionato come canali per il trasporto stimolato di specie di carico attraverso le membrane.
Contrariamente, il presente studio propone il concetto di fabbricazione attiva di nanofori in piastrelle origami.
Si segnala la formazione attiva guidata da DNAzyme di nanofori in impalcature origami e lo sblocco meccanico molecolare dei nanofori sollevando i domini a finestra coperti.
Applicando due diversi DNAzymes, viene dimostrata la fabbricazione programmata ed attivata di nanofori nelle strutture di origami.
Inoltre, si utilizza le cavità presenti nei diversi scaffold di origami come nanoambienti confinati per catalisi selettiva e specifica“.
In questa spiegazione, che non lascia dubbi sull’intenzionalità della fabbricazione della tecnica dell’origami, c’è un dettaglio fondamentale che deve essere preso seriamente in considerazione.
Tale dettaglio concerne la capacità delle cavità nei reticoli di origami del DNA di intrappolare, immobilizzare e fissare gli anticorpi (Ouyang, X.; De-Stefano, M.; Krissanaprasit, A.; Bank-Kodal, A. L.; Bech-Rosen, C.; Liu, T.; Gothelf, KV 2017), che originariamente era destinata a studi sierologici, ma che, quando applicata alla costruzione di dispositivi elettronici intra-corpo in micro/nano-scala, può raggiungere l’obiettivo di prevenire la fagocitosi e l’immobilizzazione di strutture auto-formanti.
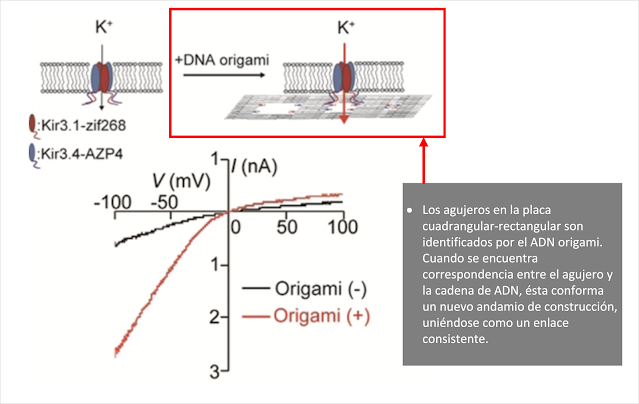
Inoltre, è stato evidenziato che questi fori svolgono un ruolo cruciale nell’interazione con altre sequenze di reticoli di origami di DNA, che possono combaciare tra loro come se fossero pezzi di Lego, aggiungendo nuove impalcature di costruzione (Kurokawa, T. ; Kiyonaka, S.; Nakata, E.; Endo, M.; Koyama, S.; Mori, E.; Mori, Y. 2018), come nella Figura 7.
Un’ulteriore applicazione menzionata da Wang e dal suo team per i fori è quella di fungere da canali o pori attraverso la piastra o la struttura origami di DNA per lo sviluppo di biosensori, come confermato da (Seifert, A.; Göpfrich, K.; Burns, JR.; Fertig, N.; Keyser, UF.; Howorka, S., 2015 | Burns, JR.; Seifert, A.; Fertig, N.; Howorka, S., 2016).
Si afferma infatti che “i nanopori che attraversano la membrana a partire dal DNA piegato sono un esempio recente di nanostrutture biomimetiche ingegnerizzate che possono aprire applicazioni nei biosensori, nella consegna dei farmaci e nella nanofluidica…Si stabilisce che i pori del DNA presentano due stati di conduttanza dipendenti dalla tensione.
Basse tensioni transmembrana favoriscono un livello stabile di alta conduttanza, che corrisponde a un poro del DNA non ostruito.
La larghezza interna prevista del canale aperto è confermata misurando la variazione di conduttanza in funzione della dimensione del poli (etilenglicole) (PEG), pertanto si presume che i PEG più piccoli entrino nel poro“.
Questo elemento corrisponde non solo a uno dei componenti elencati negli eccipienti del cosiddetto “vaccino” Pfizer, ma anche alla conduttanza richiesta per i componenti della nano-rete orientata al corpo umano (Yang, J.).; Ma, M. ; Li, L.; Zhang, Y.; Huang, W.; Dong, X. 2014 | Abbasi, Q. H.; Yang, K.; Chopra, N.; Jornet, J. M.; Abuali, N. A.; Qaraqe, K. A.; Alomainy, A. 2016 | Oukhatar, A.; Bakhouya, M.; El Ouadghiri, D. 2021).
Bibliografia
1.Abbasi, QH; Yang, K.; Chopra, N.; Jornet, JM; Abuali, NA; Qaraqe, KA; Alomania, A. (2016). Nano-communication for biomedical applications : A review on the state-of-the-art from physical layers to novel networking concepts. IEEE Access, 4, pp. 3920-3935. https://doi.org/10.1109/ACCESS.2016.2593582
2.Burns, JR; Seifert, A.; Fertig, N.; Howorka, S. (2016). A biomimetic DNA-based channel for the ligand-controlled transport of charged molecular cargo across a biological membrane. Nature nanotechnology, 11 (2), pp. 152-156. https://doi.org/10.1038/nnano.2015.279
3.Burns, MA; Mastrangelo, CH; Sammarco, TS; Man, FP; Webster, JR; Johnson, BN; Burke, DT (1996). Microfabricated structures for integrated DNA analysis. Proceedings of the National Academy of Sciences, 93 (11), pp. 5556-5561. https://doi.org/10.1073/pnas.93.11.5556
4.Catania, V.; Mineo, A.; Monteleone, S.; Patti, D. (2014). Distributed topology discovery in self-assembled nano network-on-chip. Computers & Electrical Engineering, 40 (8), pp. 292-306. https://doi.org/10.1016/j.compelecita.2014.09.003
5.Chen, Y.; Pepin, A. (2001). Nanofabrication : Conventional and nonconventional methods. Electrophoresis, 22 (2), pp. 187-207. https://doi.org/10.1002/1522-2683(200101)22:2%3C187::AID-ELPS187%3E3.0.CO;2-0
6.Endo, M.; Sugiyama, H. (2014). Single-molecule imaging of dynamic motions of biomolecules in DNA origami nanostructures using high-speed atomic force microscopy. Accounts of chemical research, 47 (6), pp. 1645-1653. https://doi.org/10.1021/ar400299m
7.Esener, SC; Hartmann, DM; Heller, MJ; Cable, JM (1998). DNA-assisted microassembly : a hetrogeneous integration technology for optoelectronics. En: Heterogeneous Integration: Systems on a Chip: A Critical Review (Vol. 10292, p. 1029208). International Society for Optics and Photonics. https://doi.org/0.1117/12.300616
8.Gabrys, PA; Seo, SE; Wang, MX; Oh, E.; Macfarlane, RJ; Mirkin, CA (2018). Lattice mismatch in crystalline nanoparticle thin films. Nano letters, 18 (1), pp. 579-585. https://doi.org/10.1021/acs.nanolett.7b04737
9.Galal, A.; Hesselbach, X. (2018). Nano-networks communication architecture : Modeling and functions. Nano Communication Networks, 17, pp. 45-62. https://doi.org/10.1016/j.nancom.2018.07.001
10.Galal, A.; Hesselbach, X. (2020). Probability-based path discovery protocol for electromagnetic nano-networks. Computer Networks, 174, 107246. https://doi.org/10.1016/j.comnet.2020.107246
11.Gu, H.; Chao, J.; Xiao, SJ; Seeman, NC (2010). A proximity-based programmable DNA nanoscale assembly line. Nature, 465 (7295), pp. 202-205. https://doi.org/10.1038/nature09026
12.Hong, F.; Zhang, F.; Liu, Y.; Yan, H. (2017). DNA origami : scaffolds for creating higher order structures. Chemical reviews, 117 (20), pp. 12584-12640. https://doi.org/10.1021/acs.chemrev.6b00825
13.Keren, K.; Berman, RS; Buchstab, E.; Sivan, U.; Braun, E. (2003). Transistor de efecto de campo de nanotubos de carbono con plantilla de ADN. Science, 302 (5649), pp. 1380-1382. https://doi.org/10.1126/science.1091022 | https://sci-hub.yncjkj.com/10.1126/science.1091022
14.Krahne, R.; Yacoby, A.; Shtrikman, H.; Bar-Joseph, I.; Dadosh, T.; Sperling, J. (2002). Fabrication of nanoscale gaps in integrated circuits. Applied physics letters, 81 (4), pp. 730-732. https://doi.org/10.1063/1.1495080
15.Kumar, P. (2010). Directed self-assembly : expectations and achievements. Nanoscale research letters, 5 (9), pp. 1367-1376. https://doi.org/10.1007/s11671-010-9696-9 | https://sci-hub.yncjkj.com/10.1007/s11671-010-9696-9
16.Kurokawa, T.; Kiyonaka, S.; Nakata, E.; Endo, M.; Koyama, S.; Mori, E.; Mori, Y. (2018). DNA origami scaffolds as templates for functional tetrameric Kir3 K+ channels. Angewandte Chemie International Edition, 57 (10), pp. 2586-2591. https://doi.org/10.1002/anie.201709982
17.Lewis, DJ; Zornberg, LZ; Carter, DJ; Macfarlane, RJ (2020). Single-crystal Winterbottom constructions of nanoparticle superlattices. Nature materials, 19 (7), pp. 719-724. https://doi.org/10.1038/s41563-020-0643-6
18.Liu, J.; Lu, Y. (2003). A colorimetric lead biosensor using DNAzyme-directed assembly of gold nanoparticles. Journal of the American Chemical Society, 125 (22), pp. 6642-6643. https://doi.org/10.1021/ja034775u
19.Liu, J.; Lu, Y. (2006). Fast colorimetric sensing of adenosine and cocaine based on a general sensor design involving aptamers and nanoparticles. Angewandte Chemie, 118 (1), pp. 96-100. https://doi.org/10.1002/ange.200502589
20.Liu, J.; Wei, J.; Yang, Z. (2021). Building ordered nanoparticle assemblies inspired by atomic epitaxy. Physical Chemistry Chemical Physics, 23 (36), pp. 20028-20037. https://doi.org/10.1039/D1CP02373J | https://sci-hub.yncjkj.com/10.1039/D1CP02373J
21.Lund, K.; Manzo, AJ; Dabby, N.; Michelotti, N.; Johnson-Buck, A.; Nangreave, J.; Yan, H. (2010). Molecular robots guided by prescriptive landscapes. Nature, 465 (7295), pp. 206-210. https://doi.org/10.1038/nature09012
22.McMillan, RA; Pavola, CD; Howard, J.; Chan, SL; Zaluzec, NJ ; Trent, JD (2002). Ordered nanoparticle arrays formed on engineered chaperonin protein templates. Nature materials, 1 (4), pp. 247-252. https://doi.org/10.1038/nmat775
23.Nykypanchuk, D.; Maye, MM; Van-Der-Lelie, D.; Gang, O. (2008). DNA-guided crystallization of colloidal nanoparticles. Nature, 451 (7178), pp. 549-552. https://doi.org/10.1038/nature06560
24.Omabegho, T.; Sha, R.; Seeman, NC (2009). A bipedal DNA Brownian motor with coordinated legs. Science, 324 (5923), pp. 67-71. https://doi.org/10.1126/science.1170336
25.Oukhatar, A.; Bakhouya, M.; El Ouadghiri, D. (2021). Electromagnetic-Based Wireless Nano-Sensors Network : Architectures and Applications. J. Commun, 16 (1), 8. https://www.researchgate.net/profile/Driss-El-Ouadghiri/publication/348355577…/Electromagnetic-Based-Wireless-Nano-Sensors-Network-Architectures -e-Applicazioni.pdf
26.Ouyang, X.; De-Stefano, M.; Krissanaprasit, A.; Banca-Kodal, AL; Bech-Rosen, C.; Liu, T.; Gothelf, KV (2017). Docking of antibodies into the cavities of DNA origami structures. Angewandte Chemie, 129 (46), pp. 14615-14619. https://doi.org/10.1002/ange.201706765
27.Rothemund, PW (2006). Folding DNA to create nanoscale shapes and patterns. Nature, 440 (7082), pp. 297-302. https://doi.org/10.1038/nature04586
28.Sarlangue, G.; Devilleger, J.; Trillaud, P.; Fouchet, S.; Taillasson, L.; Catteau, G. (2021). Objectivation de l’existence d’adresse MAC détectables sur la plage de fréquence Bluetooth suite à une inoculation de thérapie antigénique COVID et de test PCR de détection COVIDhttps://ln5.sync.com/dl/195df4a10/5ab9apq6-q5vgawam-vgr3ktt9-7zr985rh/view/default/2451906512011
29.Seifert, A.; Göpfrich, K.; Burns, JR; Fertig, N.; Keyser, UF; Howorka, S. (2015). Bilayer-spanning DNA nanopores with voltage-switching between open and closed state. ACS nano, 9 (2), pp. 1117-1126. https://doi.org/10.1021/nn5039433
30.Shen, J.; Sole, O.; Liu, D.; Schaus, T.; Yin, P. (2021). Three-dimensional nanolithography guided by DNA modular epitaxy. Nature Materials, 20 (5), pp. 683-690. https://doi.org/10.1038/s41563-021-00930-7
31.Taton, TA; Mirkin, California; Letsinger, RL (2000). Scanometric DNA array detection with nanoparticle probes. Science, 289 (5485), pp. 1757-1760. https://doi.org/10.1126/science.289.5485.1757
32.Wang, J.; Yue, L.; Li, Z.; Zhang, J.; Tian, H.; Willner, I. (2019). Active generation of nanoholes in DNA origami scaffolds for programmed catalysis in nanocavities. Nature communications, 10 (1), pp. 1-10. https://doi.org/10.1038/s41467-019-12933-9 | https://www.researchgate.net/publication/336927899_Active_generation_of_nanoholes_in_DNA_origami_scaffolds_for_programmed_catalysis_in_nanocavities
33.Wang, MX; Seo, SE; Gabrys, PA; Fleischman, D.; Lee, B.; Kim, Y.; Mirkin, CA ; (2017). Epitaxy : Programmable atom equivalents versus atoms. ACS nano (1), pp. 180-185. https://doi.org/10.1021/acsnano.6b06584
34.Yang, J.; Ma, M.; Li, L.; Zhang, Y.; Huang, W.; Dong, X. (2014). Graphene nanomesh : new versatile materials. Nanoscale, 6 (22), pp. 13301-13313. https://doi.org/10.1039/C4NR04584J | https://sci-hub.yncjkj.com/10.1039/c4nr04584j
35.Zhang, R.; Yang, K.; Abbasi, QH; Qaraqe, KA; Alomania, A. (2017). Analytical characterisation of the terahertz in-vivo nano-network in the presence of interference based on TS-OOK communication scheme. IEEE Access, 5, pp. 10172-10181. https://doi.org/10.1109/ACCESS.2017.2713459







![Bambini pandemici Screenshot 2021-09-25 at 12-04-03 Rêve was 2 days old in the NICU, less than 2 lb and can hold here up straight to look at […]](https://it.nuda-verita.com/wp-content/uploads/2021/09/Screenshot-2021-09-25-at-12-04-03-Reve-was-2-days-old-in-the-NICU-less-than-2-lb-and-can-hold-here-up-straight-to-look-at--150x150.png)